最近数年,随着高通量测序技术的发展,人们对环状RNA(circular RNA,circRNA)的认识不断增加,circRNA很多功能被发现。最近出版的nature structural & molecular biology,报道了中科大、华中师范大学等处关于circRNA在细胞核中转录调控功能最新研究。
通过RNA测序和分析,人们已在动物细胞中已发现很多circRNA,发现circRNA经常表现在发育过程中表现出时期和组织特异性,表明其可能具有特殊调控功能。
本项研究中,研究者希望鉴定出一些与RNA Pol II互作的ncRNA,他们在HeLa细胞中用Pol II抗体进行了CLIP-Seq实验,共鉴定出111个circRNA,并通过RIP、northern blot和qPCR对其中表达量最高的两个circRNA:circEIF3J和circPAIP2进行了验证,结果发现这两个circRNA中,外显子 之间都保留有内含子序列,研究者将这类circRNA命名为EIciRNA(Exon-intron circular RNAs)。对于EIciRNA产生原因,研究者通过构建不同circEIF3J和circPAIP2基因序列表达质粒证明,EIciRNA会受到其所对 应基因旁侧序列(flanking sequence)中的Alu元件和反向重复序列刺激,发生过表达。
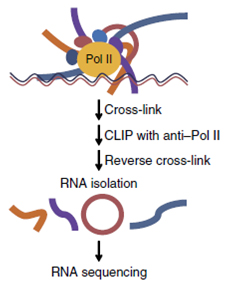
Pol II CLIP-Seq示意图
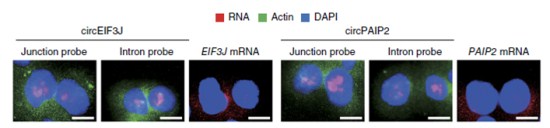
circEIF3J和circPAIP2 FISH结果
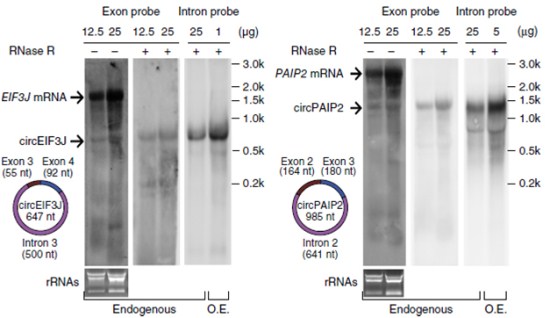
ircEIF3J和circPAIP2 northern blot结果表明,两个circRNA中都含有内含子序列
在进一步EIciRNA功能研究中,研究者首先进行了RNA-DNA双FISH实验,结果表明,两个EIciRNA与其所对应的parental gene共定位于核内,再加上两个EIciRNA与Pol II的互作关系,表明EIciRNA可能对其parental gene具有调控功能。在HEK293细胞中进行GRO-Seq发现,当使用反义核酸(antisense oligonucleotides,ASO)分别沉默circEIF3J和circPAIP,会造成EIF3J和PAIP2转录下降,这些数据证实,EIciRNA会通过与Pol II互作,反式调控其parental gene表达。
接下来研究者分别通过RNA pull down和ChIRP实验分析了与两个EIciRNA互作的蛋白及DNA。在RNA pull down实验中,研究者不单单发现了Pol II,还发现了U1A和U1C snRNP(small nuclear RNA binding protein),EIciRNA和U1 snRNA双RNA FISH实验表明circEIF3J和circPAIP与U1 snRNA在核内互作,而ChIRP实验则表明两个EIciRNA与Pol II、U1 snRNP共同结合于其parental gene转录起始位点上游300bp处,当U1 snRNA被沉默,CLIP及ChIP实验表明,EIciRNA同Pol II、parental gene启动子区域的互作会减少;如果用抑制物封闭U1 snRNA在EIciRNA的结合位点,也会产生相同结果。这些实验数据表明,两个EIciRNA会与U1 snRNP、Pol II互作,结合于其parental gene的转录起始位点处,对基因转录进行调控,且U1 snRNP的结合是必需的。
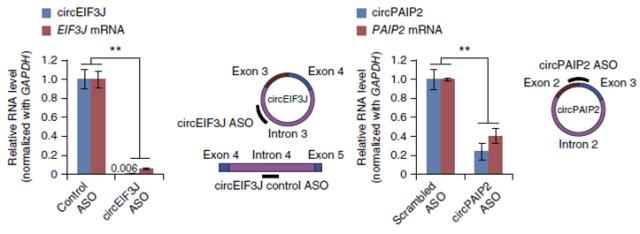
使用ASO沉默两个EIciRNA后,其所对应parental gene转录同样受到抑制
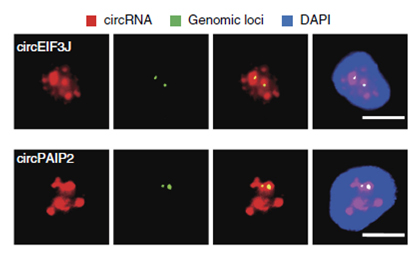
EIciRNA与其parental gene进行RNA-DNA双FISH结果,从图中可以看出,EIciRNA与其parental gene共定位于核内
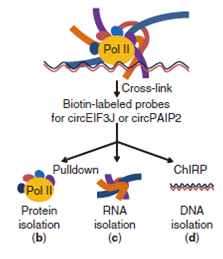
EIciRNA ChIRP示意图
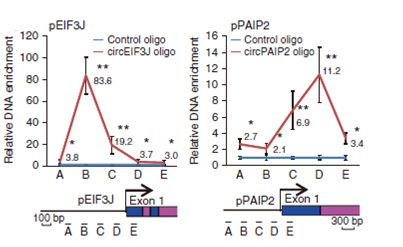
EIciRNA ChIRP结果
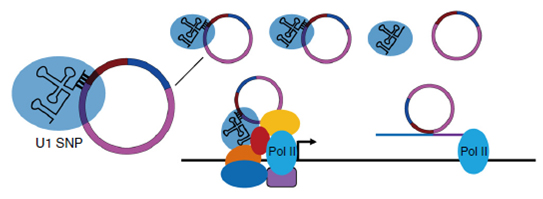
本项研究示意图 EIciRNA与U1 snRNP、Pol II互作,结合于parental gene转录起始位点上游,调控parental gene转录
本项研究,发现了EIciRNA这种circRNA的重要功能,并对EIciRNA调控机制进行了探索,表明该类型circRNA在核内基因调控过 程中扮演了重要角色。
相关文献:Li Z, Huang C, Bao C, et al. Exon-intron circular RNAs regulate transcription in the nucleus[J]. Nature structural & molecular biology, 2015, 22(3): 256-264.